
こんにちは。FIXER M&S 竹中です。シリーズ5本目になります。今回からリアルタイムのコンペの参加報告の体で書かせて頂きます。また、コード量も増えてきたことからブログ中に全量記述することはせず、代わりに私のGithubリポジトリを公開することで代えさせて頂きます。拙いコーディングですが何かのご参考になれば幸いです。
第一回→①環境設定篇
第二回→②クラス分類入門篇
第三回→③回帰分析入門篇
第四回→④lightGBMを使ってみよう篇
番外編→Azure Data Science Virtual Machineを使ってみよう
上記リンクにもありますように、今回から計算用のリソースとしてAzureも活用を始めています。Azureにはデータ分析用のプリセットがありますので、クラウドを利用した計算に興味のある方は是非上記記事もご覧ください。
1.今回のお題
今回は“Predicting Molecular Properties”というコンペに挑戦してまいりました。どのようなコンペか?を説明するために、まずは下のアニメをご覧ください。(※出展)
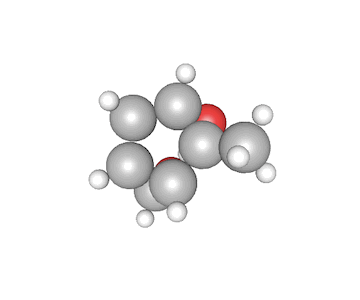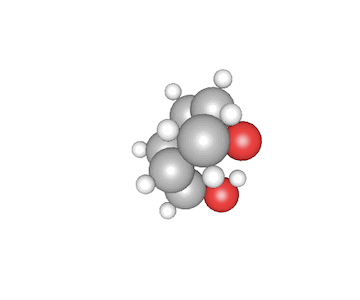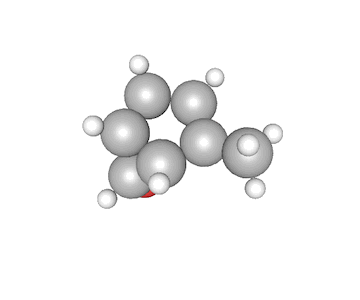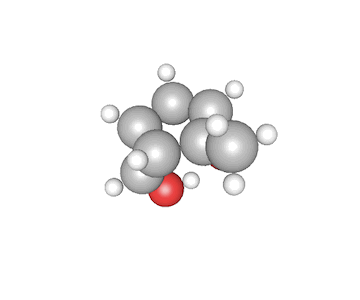
これが何か分かりますでしょうか?これはある特定の分子の構造を示しています。球体の一つ一つが原子を表しており、一つの分子は固有の構造を取ります。
原子は様々な相対位置、結合の方式を持っており、それらのパラメータから「結合定数」と呼ばれる固有の物理量を持ちます。今回のコンペでは、分子に含まれる原子の種類とxyz座標から結合定数を推定するというもので、回帰型のモデルが要求されています。
ただし、分子に含まれるすべての原子は明らかにされておらず、各2つずつの原子が表現されているのみです。
2.まずはデータを見てみよう
いつも通り、まずはデータを見てみましょう。
# サンプルからデータ型を調べる関数
def Datatype_table(df):
list_type = df.dtypes #データ型
Datatype_table = pd.concat([list_type], axis = 1)
Datatype_table_len = Datatype_table.rename(columns = {0:'データ型'})
return Datatype_table_len
Datatype_table(train_csv)
Datatype_table(structures_csv)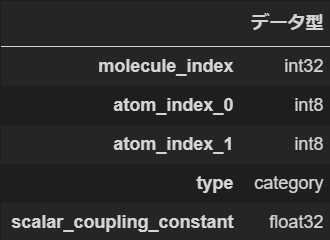
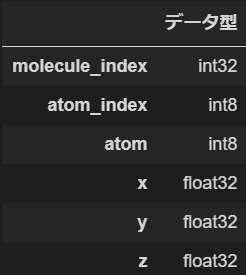
上が訓練データ、下がxyz座標のデータです。この2つをマージすることで今回の基本的なデータセットが出来上がります。テストデータはscalar_coupling_constantのカラムが落ちているだけで他は訓練データと同様の構造です。欠損データはありません。ちなみに今回訓練データが約460万レコード、テストデータが260万レコードほどあり学習負荷は相応に高めです。
大容量なデータをクラウド上で簡単に計算してみた方法はこちら。
ブログ:Azure Data Science Virtual Machines(DSVM)を使ってみよう
これだけではデータの構造が良く分かりませんのでヒストグラムを見てみましょう。縦軸が結合定数、横軸はTypeといって分子の結合の方式全8種類を示しています。また、全量のプロットだとデータ数が多すぎますのでPandasの機能で一部をランダムサンプリングしておきましょう。
train_sample = train_csv.sample(frac=0.05)
plt.scatter(train_sample["type"], train_sample["scalar_coupling_constant"])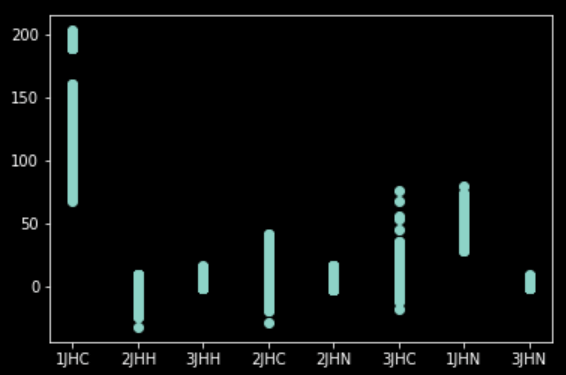
結合のタイプによって定数が違う、という傾向が見て取れますね。逆にすべてのデータを一緒にしてしまうと分布が悪くなってしまうため、今回は1JHC~3JNNの8種類のモデルを別々に学習、予測させましょう。最終結果をID付きでマージすれば提出用のデータセットが作れるはずです。
3.特徴量とデータ分割を工夫してみる
今回特徴量の作り方としては Marco Gorelli 氏のKernelを大いに参考にしています。KernelとはKaggler達が残したノートのようなもので、中にはkaggle masterと呼ばれる偉人たちのコードが掲載されていたりもします。Marco氏自身もSamsung UKでデータサイエンティストとして働くその道のプロ、とBioには書かれていました。こうして「巨人の肩に乗る」の発想で優れたアイデアを取り込み更に発展させることでコンペ自体がハイレベルになっていく、というのがKaggleの素晴らしいところかと思います。
4.モデルにフィッティングしよう
さて、データセットを用意したところでフィッティングしてみましょう。今回は8つのデータセットがあるので、モデルも別々に適用が可能です。上記分子タイプのうち、「1JHN」だけはサンプル数が全体の1%と少なく学習効率が悪いことが想定されますので、効率の高いlightGBMを利用します。その他については今回はRandomForestRegresserを利用しています。
実験的にRandomForestの方が精度が高いことが分かりましたが、ハイパーパラメータのn_estimator(簡単に言うと、吟味する決定木の数)がデフォルトだとパフォーマンスが悪いため500まで増やしています。本当は1000ならもう少し良い数字が出るのですが、estimatorを増やしてCrossValidationするととんでもない時間がかかったので多少チューニングしています。
このプロジェクトはあくまで私個人の取り組みなので、Azure代も自腹…。計算時間と精度のトレードオフはお財布と相談しました(泣)
def try_fit_predict_lgbm(train_df, test_df, index_df, savename):
X_data = train_df.drop(['scalar_coupling_constant'], axis=1).values.astype('float32')
y_data = train_df['scalar_coupling_constant'].values.astype('float32')
test_feature = test_df
X_train, X_test, y_train, y_test = train_test_split(X_data , y_data , test_size=0.2, random_state=128)
# LGBMRegressorによる予測
LGB_PARAMS = {
'objective': 'regression',
'metric': 'mae',
'verbosity': -1,
'boosting_type': 'gbdt',
'learning_rate': 0.2,
'num_leaves': 128,
'min_child_samples': 79,
'max_depth': 9,
'subsample_freq': 1,
'subsample': 0.9,
'bagging_seed': 11,
'reg_alpha': 0.1,
'reg_lambda': 0.3,
'colsample_bytree': 1.0
}
model2 = lgb.LGBMRegressor(**LGB_PARAMS, n_estimators=20000, n_jobs = -1)
print('Start Fitting')
model2.fit(X_train, y_train,
eval_set=[(X_train, y_train), (X_test, y_test)], eval_metric='mae',
verbose=500, early_stopping_rounds=1000)
print('Start Getting Mae')
prediction_rf_mae = model2.predict(X_test)
Err = mae(y_test, prediction_rf_mae)
acc_dic[savename] = Err
print('Start Predicting')
prediction_lgb = model2.predict(test_feature)
index_df['scalar_coupling_constant'] = prediction_lgb
csv_title = 'result_' + savename + '.csv'
index_df.to_csv(csv_title)
return prediction_lgb
def try_fit_predict_RandomForest(train_df, test_df, index_df, savename):
X_data = train_df.drop(['scalar_coupling_constant'], axis=1).values.astype('float32')
y_data = train_df['scalar_coupling_constant'].values.astype('float32')
test_feature = test_df
X_train, X_test, y_train, y_test = train_test_split(X_data , y_data , test_size=0.33, random_state=128)
# RandomForestRegressorによる予測
params = {'n_estimators' : [500], 'n_jobs': [-1]}
forest = RandomForestRegressor()
model = GridSearchCV(forest, params, cv = 3)
print('Start Fitting')
model.fit(X_train, y_train)
print('Start Getting Mae')
prediction_rf_mae = model.predict(X_test)
Err = mae(y_test, prediction_rf_mae)
acc_dic[savename] = Err
print('Start Predicting')
prediction_rf = model.predict(test_feature)
index_df['scalar_coupling_constant'] = prediction_rf
csv_title = 'result_' + savename + '.csv'
index_df.to_csv(csv_title)
return prediction_rf5.さて、結果は…??
さて、そんなこんなで試行錯誤し、最終的に期限までに自分が選ぶスコアを提出します。チェックマークを付けない場合は最良スコアが勝手に提出されたことになるのでうっかり忘れていても大丈夫です(ただし、コンペ中に見えているスコアはPublic Scoreといって提出データから一部をサンプリングして算出しています。最終は全量でスコアしますので、自信のあるデータを選択されることをお勧めします)
で、こちらが私の結果です!

はい、今回のスコアは2749チーム中1076位とイマイチでしたね。敗因はいくつかあると思っているのですが
- 特徴量の追い込み不足(多分一番要因としては大きい)
- モデルのチューニング不足
- 計算時間が長いことを見越したスケジューリングができていなかった
- 分子構造の知識がなかった(データに関するイメージが持てるか、前提知識があるか、は意外と大事)
辺りかと思います。あと、〆切間近になるとみんな本気モードになってKernelも活発になりましたが、業務都合もありキャッチアップできていませんでした。ここでもう少しコードをブラッシュアップできていればより上位を狙えたかもしれません。
さて、今回はリアルタイムのコンペ初挑戦ということで諸々上手く運べなかった点もありますが、過程においてAzureの使い方を覚えたりPythonもちょっとは上手くなったり学びはあったかと思います。
みんなもKaggle、やろう!!
今回は以上です。お読みいただきありがとうございました。






![Microsoft Power BI [実践] 入門 ―― BI初心者でもすぐできる! リアルタイム分析・可視化の手引きとリファレンス](/assets/img/banner-power-bi.c9bd875.png)
![Microsoft Power Apps ローコード開発[実践]入門――ノンプログラマーにやさしいアプリ開発の手引きとリファレンス](/assets/img/banner-powerplatform-2.213ebee.png)
![Microsoft PowerPlatformローコード開発[活用]入門 ――現場で使える業務アプリのレシピ集](/assets/img/banner-powerplatform-1.a01c0c2.png)


